Ein molekulares Kontrollzentrum sorgt für Ordnung
Wie werden Proteine in unseren Zellen noch während ihrer Herstellung modifiziert? Ein internationales Team aus Forschenden der Universität Konstanz, des Caltech und der ETH Zürich entschlüsselte den molekularen Mechanismus dieses Vorgangs, der für unser Leben und unsere Gesundheit essenziell ist.
Proteine gehören zu den wichtigsten molekularen Bausteinen des Lebens. Sie entstehen durch die Zusammenkettung von Aminosäuren auf Grundlage der Informationen unseres Erbguts. Dabei wird der Code unserer Gene in eine Abfolge aus Aminosäuren übersetzt. Diese „Translation“ ist jedoch nur der erste Schritt. Häufig werden Proteine noch während sie ihre zelluläre Produktionsstätte – das Ribosom – verlassen durch spezielle Enzyme modifiziert. Erst danach können die Proteine ihre vielfältigen biologischen Funktionen erfüllen.
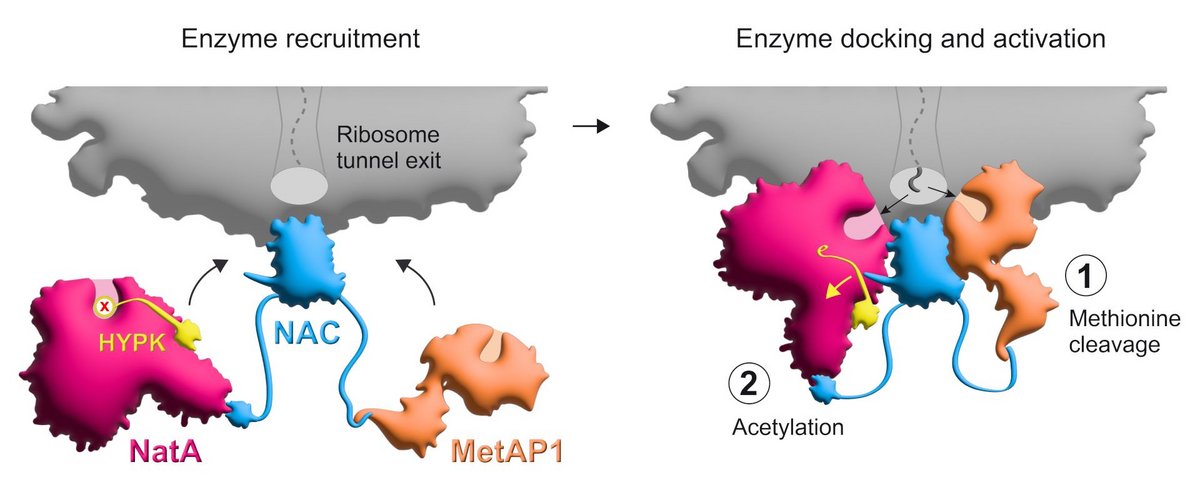
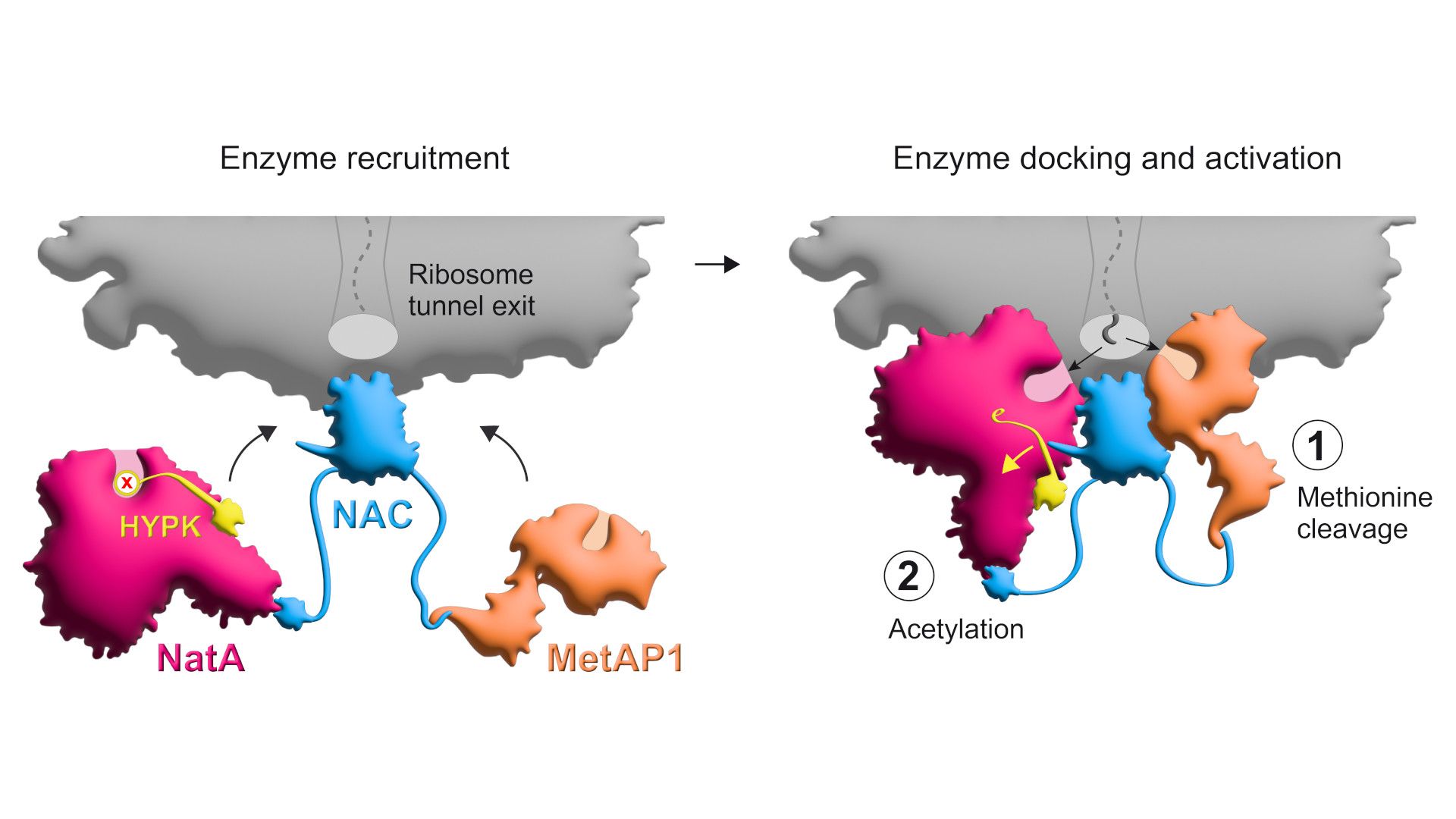
Wie diese Enzyme bei der Modifikation von Proteinen am Ribosom zusammenarbeiten und wie ihre Aktivität dabei reguliert und koordiniert wird, war bisher nur bruchstückhaft bekannt. Einem internationalen Team aus Forschenden der Universität Konstanz, des California Institute of Technology (Caltech) und der ETH Zürich ist es nun gelungen, den komplexen molekularen Mechanismus für zwei aufeinanderfolgende Proteinmodifikationen zu entschlüsseln, denen bei Säugetieren etwa 40 Prozent aller Proteine unterliegen. Koordiniert wird dieser überlebenswichtige Vorgang durch einen Proteinkomplex namens NAC (kurz für „nascent polypeptide-associated complex“). Die Ergebnisse wurden in der Fachzeitschrift Nature publiziert.
Für eine normale Zellfunktion elementar
Die Modifikationen, um die es sich konkret handelt, sind die Abspaltung der Aminosäure Methionin vom wachsenden Protein, gefolgt von dem Anhängen einer sogenannten Acetylgruppe an das verbleibende Ende. Beides geschieht am ribosomalen Tunnel, also an dem Ort, wo die Proteine während ihrer Herstellung das Ribosom als wachsende Kette aus Aminosäuren verlassen. Diese Modifikationen sind für einen Großteil unserer Proteine absolut essenziell, denn sie beeinflussen gleich mehrere Proteineigenschaften – wie die dreidimensionale Faltung, die Lebensdauer oder die Interaktion mit anderen Proteinen – und damit die eigentliche Funktion der Proteine.
„Eine Fehlregulation der an der Proteinmodifikation beteiligten Vorgänge kann äußerst negative Auswirkungen für den Organismus haben und wird zum Beispiel mit Entwicklungsstörungen oder Krankheiten wie Krebs und Parkinson in Verbindung gebracht“, erklärt Elke Deuerling, Professorin am Fachbereich Biologie der Universität Konstanz. Sie und ihre Konstanzer Kollegen Martin Gamerdinger und Laurenz Rabl waren maßgeblich an der aktuellen Studie beteiligt.
Der Mechanismus im Detail
Damit die Abspaltung von Methionin und die anschließende Acetylierung reibungslos ablaufen können, steht nur ein kurzes Zeitfenster zur Verfügung. In ihm müssen gleich mehrere Enzyme zielgenau an den Ort des Geschehens gebracht und reguliert werden: MetAP1, das die Abspaltung von Methionin bewirkt, und NatA für die darauffolgende Acetylierung. NatA wird jedoch im Normalfall von einem Inhibitorprotein gebunden und dadurch in seiner Funktion gehemmt: HYPK. Durch die Kombination von biochemischen, strukturellen und in vivo Experimenten ist es den Konstanzer Forschenden und ihren Kolleg*innen aus den USA und der Schweiz nun gelungen, die Steuerung dieses komplexen Vorgangs und die Interaktion der beteiligten Makromoleküle umfänglich aufzuklären.
Der Proteinkomplex NAC nimmt dabei eine zentrale Rolle ein: NAC sitzt über dem Ausgang des ribosomalen Tunnels, aus dem die neu synthetisierten Proteine herauskommen. Dort rekrutiert es die für die Abspaltung von Methionin und die anschließende Acetylierung notwendigen Enzyme und positioniert diese mit ihren biochemisch aktiven Bereichen an genau den richtigen Stellen in der Nähe des Tunnels, sodass sie Zugang zum entstehenden Protein erhalten. „Zusätzlich bewirkt NAC, dass NatA seinen inhibitorischen Kontakt zu HYPK verliert. Dadurch wird sichergestellt, dass die Funktion von NatA nur am Ribosom aktiviert wird, wo es dann die gewünschte Acetylierung vermitteln kann“, sagt Shu-ou Shan, Altair-Professorin für Chemie und Executive Officer für Biochemie und molekulare Biophysik am Caltech.
NAC: ein allgemeines molekulares Kontrollzentrum?
Aus vorherigen Studien gibt es Hinweise darauf, dass NAC neben den Enzymen MetAP1 und NatA auch noch weitere Faktoren zum ribosomalen Tunnel rekrutiert. „Wir gehen inzwischen davon aus, dass NAC die Funktion eines noch umfangreicheren molekularen Kontrollzentrums hat. Es sorgt dafür, dass die entstehenden Proteine beim Verlassen des Ribosoms je nach Anforderung Zugang zu unterschiedlichen Komponenten des biochemischen Werkzeugkastens der Zelle bekommen“, veranschaulicht Gamerdinger die allgemeine Bedeutung von NAC.
Die aktuelle Studie zeigt, wie NAC diese wichtige Funktion im konkreten Fall erfüllt. Dieses neue Wissen über die komplexen molekularen Vorgänge in der Zelle erlaubt es uns, die biochemischen Grundlagen unseres Lebens besser zu verstehen. Darüber hinaus wird es auch zu einem besseren Verständnis davon beitragen, wie Fehlregulationen der an der Modifikation von Proteinen beteiligten Komponenten zu der Entstehung von Krankheiten führen, und könnte so auf lange Sicht Basis für die Entwicklung neuer therapeutischer Ansätze in der Medizin sein.
Faktenübersicht:
- Originalpublikation: A. Lentzsch, D. Yudin, M. Gamerdinger, S. Chandrasekar, L. Rabl, A. Scaiola, E. Deuerling, N. Ban & S. Shan (2024) NAC guides a ribosomal multienzyme complex for nascent protein processing. Nature; doi: 10.1038/s41586-024-07846-7
- Ein internationales Team aus Forschenden der Universität Konstanz, des California Institute of Technology und der ETH Zürich entschlüsselte einen molekularen Mechanismus der kotranslationalen Modifikation von Proteinen bei Eukaryoten.
- Seitens der Universität Konstanz waren Prof. Elke Deuerling, Dr. Martin Gamerdinger und Laurenz Rabl vom Fachbereich Biologie an der Studie beteiligt.
- Förderung: National Institutes of Health (NIH), National Science Foundation (NSF), Deutsche Forschungsgemeinschaft (DFG), Schweizerischer Nationalfonds (SNF) und Europäischer Forschungsrat.